在多细胞生物中,不同细胞在空间上有序排列形成了具有功能的组织和器官。邻近的细胞之间通过直接接触或者分泌可扩散的分子来完成物质交换和信息交流。为了研究细胞的空间组成,研究人员已开发了多种邻近标记技术用于对特定细胞的邻近细胞进行标记、分离和组学分析,例如LIPSTIC[1]、EXCELL[2]、FucoID[3]、μMAP[4]、PhoTag[5]及PhoXCELL[6]等。然而,受限于外源酶的作用范围或者活性中间体的半衰期,这些方法的标记半径多在纳米级别,标记范围局限于细胞接触的界面,限制了它们用于研究较弱的细胞相互作用。此外,细胞外基质中的大量蛋白容易淬灭标记,使得在组织中原位标记和研究细胞的空间组成极具挑战性。
为了解决上述问题,澳门新甫京娱乐娱城平台、北大-清华生命科学联合中心及澳门新甫京娱乐娱城平台合成与功能生物分子中心陈兴课题组于2023年5月27日在Science Advances期刊上在线发表了题为Cellular-scale proximity labeling for recording cell spatial organization in mouse tissues的研究论文,报道了一种新型的邻近细胞标记技术:QMID(quinone-methide-assisted identification of cell spatial organization)。QMID技术主要依赖于作者开发的被保护的亚甲基苯醌(quinone methide,QM)前体探针分子和相应的脱保护酶。通过在特定细胞表面展示脱保护酶并对探针分子进行脱保护,从而生成强亲电性的QM。原位生成的QM迅速与邻近细胞表面的蛋白质等亲核分子发生加成反应,实现对邻近细胞的共价标记。
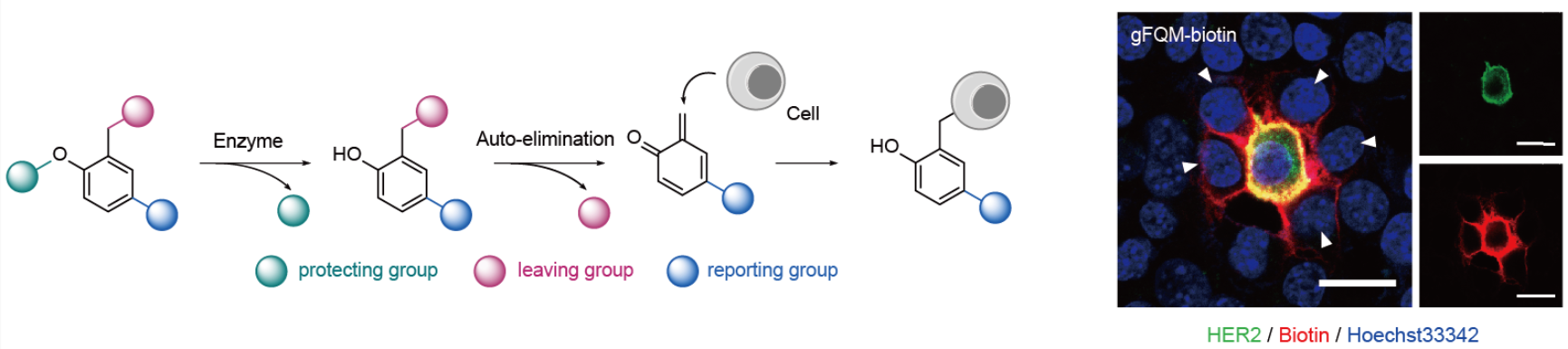
作者合成了可被半乳糖苷酶βGal激活的探针gFQM-biotin和gFMeQM-biotin。利用βGal与ZHER(HER2的纳米抗体)的嵌合蛋白ZHER-βGal选择性地在HER2阳性细胞表面展示βGal,作者证明QMID可以有效地标记HER2阳性细胞的邻近细胞。被标记后的细胞可以使用流式细胞仪或共聚焦荧光成像进行分析。
作者随后利用这一技术探究了癌细胞与巨噬细胞的相互作用。利用QMID,作者将癌细胞邻近的巨噬细胞进行了标记、分离和RNA测序。通过与单独培养的巨噬细胞的基因表达进行比较,作者发现巨噬细胞中许多基因表达与巨噬细胞和癌细胞的空间距离相关,并鉴定到PD-L1及IDO1等一系列基因表达在巨噬细胞中受到了邻近癌细胞的激活。
最后,作者将QMID应用于活体组织切片,并探究了小鼠脾脏中不同T细胞亚群的邻近细胞组成。通过生物素修饰的抗体及βGal修饰的链霉亲和素将βGal展示至小鼠脾脏切片中CD4+和CD8+T细胞的表面后,作者使用QMID对其邻近细胞进行标记和分离,结合单细胞RNA测序技术成功实现了对小鼠脾脏中两类T细胞的邻近细胞的系统鉴定与分析。QMID为研究复杂生物体系中细胞的空间组成及相互作用提供了全新的化学工具。
陈兴教授为该论文的通讯作者,博士研究生张旭和唐麒为该论文的共同第一作者。孙嘉禹、郭怡兰、张绍然、梁舒瑜、戴鹏等为该工作做出了重要贡献。该工作得到了国家自然科学基金委、科技部、北京分子科学国家研究中心以及北大-清华生命科学联合中心的资助。
[1] G. Pasqual, A. Chudnovskiy, J. M. J. Tas, M. Agudelo, L. D. Schweitzer, A. Cui, N. Hacohen, G. D. Victora, Nature 2018, 553, 496–500.
[2] Y. Ge, L. Chen, S. Liu, J. Zhao, H. Zhang, P. R. Chen, J. Am. Chem. Soc.2019, 141, 1833–1837.
[3] Z. Liu, J. P. Li, M. Chen, M. Wu, Y. Shi, W. Li, J. R. Teijaro, P. Wu, Cell2020, 183, 1117-1133.
[4] J. B. Geri, J. V. Oakley, T. Reyes-Robles, T. Wang, S. J. McCarver, C. H. White, F. P. Rodriguez-Rivera, D. L. Parker, E. C. Hett, O. O. Fadeyi, R. C. Oslund, D. W. C. MacMillan, Science.2020, 367, 1091–1097.
[5] R. C. Oslund, T. Reyes-Robles, C. H. White, J. H. Tomlinson, K. A. Crotty, E. P. Bowman, D. Chang, V. M. Peterson, L. Li, S. Frutos, M. Vila-Perelló, D. Vlerick, K. Cromie, D. H. Perlman, S. Ingale, S. D. O. Hara, L. R. Roberts, G. Piizzi, E. C. Hett, D. J. Hazuda, O. O. Fadeyi, Nat. Chem. Biol.2022, 18, 850–858.
[6] H. Liu, H. Luo, Q. Xue, S. Qin, S. Qiu, S. Liu, J. Lin, J. P. Li, P. R. Chen, J. Am. Chem. Soc.2022, 144, 5517–5526.