生物体中几乎所有的细胞都具有相同的基因组,而不同的细胞类型和功能则由不同的基因表达、表观遗传修饰和翻译后修饰等所决定。解析特定器官或组织中特定细胞的生物大分子图谱对于探究发育、细胞间通讯以及疾病的发生发展等都具有重要意义。因此,开发细胞选择性的生物大分子标记方法,近年来受到了科学家们的广泛关注1。通过基因编码的方法,人们在活体动物中实现了蛋白质的组织特异性和细胞选择性标记和分析2,3。然而,糖质(glycan)作为另外一种主要的生物大分子,尚无法通过基因编码的方式,实现活体中的细胞选择性标记。
糖质以寡糖、多糖、糖蛋白、糖脂等形式直接参与细胞的分化增殖、免疫调节、信号转导、细胞迁移等重要的生命活动,对其进行在体标记和分析一直是领域内的一个难点4。其中,基于生物正交化学的代谢糖质标记(metabolic glycan labeling)技术已经成为了最主要的工具之一。经过二十多年的发展,目前已有数十种非天然糖分子可用以在活细胞和活体中标记糖质。然而,非天然糖在活体中并不具备器官或细胞特异性,无法实现精准的细胞选择性标记,阐释特定细胞群体中糖质所发挥的生物学功能。澳门新甫京娱乐娱城平台化学与分子工程学院、北大-清华生命科学联合中心陈兴课题组一直致力于解决这个问题,此前开发了基于靶向性脂质体的非天然糖代谢标记技术,实现了肿瘤组织和脑部的糖质标记5,6。同时,他们意识到,基因编码技术可以在活体中实现更加精准的细胞选择性。为了实现这一目标,继续推进代谢糖质标记技术的应用,2022年5月5日,该课题组在Nature Chemical Biology上发表了题为Cell-type-specific labeling and profiling of glycans in living mice的论文,报道了一种可基因编码的代谢糖质标记技术(GeMGL)。该技术将“凸凹互补(bump and hole)”的化学遗传学策略与代谢糖质标记方法相结合,利用非天然糖1,3-Pr2GlcNAl(Bump)及其匹配的焦磷酸酶突变体AGX2F383G(Hole)的正交组合,在活体动物上实现了细胞选择性糖质标记和分析。

他们从一个具有低标记效率的非天然糖—乙酰胺基葡萄糖的叠氮类似物GlcNAz出发,确认了其代谢通路中的焦磷酸酶AGX是限速酶,将其过表达可以增强代谢强度。他们随即想到,增大非天然基团并对AGX酶进行突变,可能可以开发出凹凸对。于是,他们采用了炔基修饰的乙酰胺基葡萄糖GlcNAl和焦磷酸酶突变体AGX2F383G,通过体外和细胞实验证明了GlcNAl的代谢完全依赖焦磷酸酶突变体AGX2F383G。接着,在多细胞共培养体系和小鼠移植瘤模型中,证明了GeMGL策略的可行性。
基于此,他们将该策略拓展到了转基因小鼠中。他们首先利用心肌细胞特异的启动子α-MHC实现了AGX2F383G在小鼠心肌细胞中的特异性表达,然后腹腔注射非天然糖1,3-Pr2GlcNAl,实现了非天然糖分子在小鼠心肌细胞中的特异性代谢。从各组织标记结果来看,GeMGL策略展现出严格的心肌细胞选择性。结合定量蛋白质组学方法,在小鼠心肌细胞中鉴定到582个O-GlcNAc修饰蛋白。分析发现,心肌细胞中许多糖酵解、TCA循环和氧化磷酸化途径相关蛋白都具有O-GlcNAc糖基化修饰,表明O-GlcNAc糖基化修饰可能在心肌细胞的线粒体能量代谢过程中发挥重要功能。
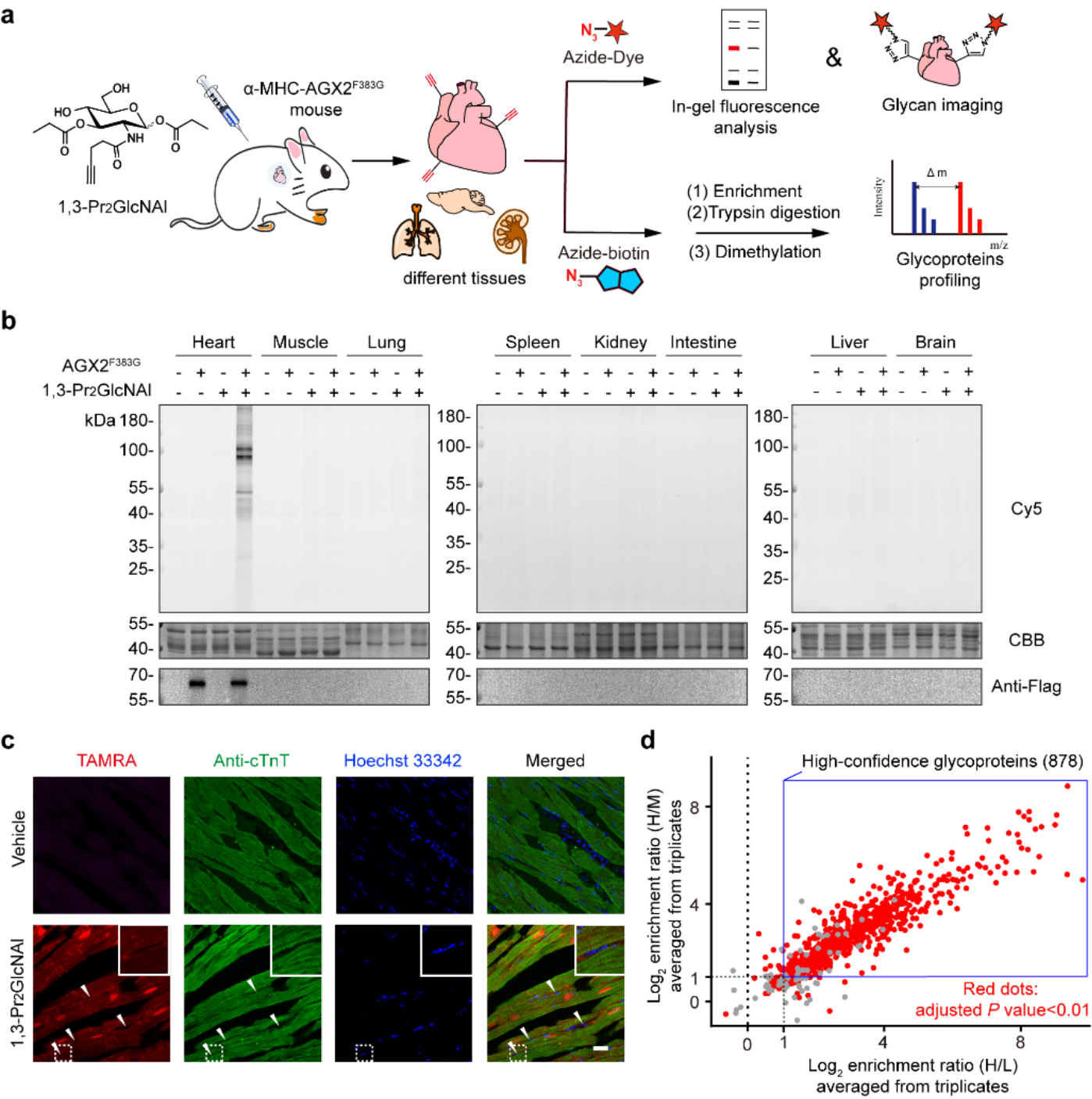
陈兴课题组长期致力于糖化学和糖生物学研究,糖质标记和分析是其研究重点之一。该工作提供了一种可基因编码的细胞特异性糖质标记技术GeMGL,为在活体层面研究糖质在特定细胞类型中的生物学功能提供了一种便利、有效的工具。该技术有望被推广到更为复杂的神经系统中,并在相关疾病模型中探究糖基化与神经发育、神经退行性疾病等的关系。
澳门新甫京娱乐娱城平台、北大-清华生命科学联合中心的陈兴教授为该论文的通讯作者,学院已毕业博士生范欣琦和生命科学联合中心博士生宋其涛为论文的共同第一作者。该研究工作得到了国家自然科学基金委、科技部、北大-清华生命科学联合中心和北京分子科学国家研究中心的资助。
原文连接:https://www.nature.com/articles/s41589-022-01016-4.
参考文献
1. Xie, R., Hong, S. & Chen, X. Cell-selective metabolic labeling of biomolecules with bioorthogonal functionalities. Curr Opin Chem Biol 17, 747–752 (2013).
2. Alvarez-Castelao, B. et al. Cell-type-specific metabolic labeling of nascent proteomes in vivo. Nat Biotechnol 35, 1196–1201 (2017).
3. Krogager, T. P. et al. Labeling and identifying cell-specific proteomes in the mouse brain. Nat Biotechnol 36, 156–159 (2018).
4. Cheng, B., Tang, Q., Zhang, C. & Chen, X. Glycan Labeling and Analysis in Cells and In Vivo. Annu Rev Anal Chem 14, 363–387 (2021).
5. Xie, R. et al. Targeted Imaging and Proteomic Analysis of Tumor‐Associated Glycans in Living Animals. Angewandte Chemie Int Ed 53, 14082–14086 (2014).
6. Xie, R. et al. In vivo metabolic labeling of sialoglycans in the mouse brain by using a liposome-assisted bioorthogonal reporter strategy. Proc National Acad Sci 113, 5173–5178 (2016).